안녕하세요.
이번 글에서는 의료분야에서 보편적으로 많이 사용되는 segmentation 모델인 UNet에 대해서 소개해드리려고 합니다.
Conference: MICCAI 2015
Paper title: U-Net: Convolutional Networks for Biomedical Image Segmentation
Authors: Olaf Ronneberger, Philipp Fischer, Thomas Brox
ISBI cell tracking challenge: First prize
https://arxiv.org/abs/1505.04597
U-Net: Convolutional Networks for Biomedical Image Segmentation
There is large consent that successful training of deep networks requires many thousand annotated training samples. In this paper, we present a network and training strategy that relies on the strong use of data augmentation to use the available annotated
arxiv.org
지난 글("1.FCN")에서 설명한 FCN 모델 다음으로 가장 주목받은 segmentation 모델이 UNet 입니다.
UNet은 biomedical image 분야에 특화시킨 FCN을 기반 segmenation 모델입니다.
참고로 UNet 논문에서 나오는 대부분의 용어는 FCN 논문에서 나옵니다. 그래서 중간 중간에 FCN 글을 참조해달라는 글을 많이 적었는데, 해당 FCN 글은 아래 링크를 참고해주세요
(↓↓↓ FCN 글 참조 ↓↓↓)
https://89douner.tistory.com/296?category=878997
실제로 많은 의료분야에서 UNet을 베이스 모델로 하여 병변들을 segmentation 하고 있습니다.
그러므로 의료분야에서 딥러닝 기반 segmentation 모델을 이용해보려는 분들은 UNet 구조를 이해하고 UNet논문에서 언급하고 있는 여러 용어들에 친숙해 질 필요가 있습니다.
그럼 지금부터 UNet 논문을 리뷰해보도록 하겠습니다.

0. Abstract

- UNet에서 강조하고 있는 부분 중 하나는 강력한 data augmentation을 이용했다는 점입니다.
- "contracting path to capture context and a symmetric expanding path"라는 문장은 아래와 "그림1"을 그대로 설명한 문장입니다. 아래 "그림1"을 보면 contracting path를 통해 context 정보(=features from almost last layers)를 구하고, contracting path 부분과 symmetric 한 expanding path를 통해 다시 원본 이미지 크기에 맞도록 (upsampling 후) segmentation (for precise localization) 을 진행하는 작업이 수행됩니다 (사실 이 부분은 FCN과 거의 다른 부분이 없습니다.)
- FCN과 UNet의 차별점은 아래 "그림1"에서 같은 층(ex: contracting path 568×568×64 & expanding path 392×392×128)끼리 이어붙여주는 연산의 유무입니다. (좀 더 자세한 설명은 UNet 구조를 설명하는 부분에서 자세히 다루도록 하겠습니다.
- "segmentation of neuronal structures in electron microscopic stacks" → "segmentation for stacks of serial images generated by EM (Electron Microscopy(=전자 현미경)).

- UNet 모델은 ISBI cell tracking challenge 2015에 참가하여 주목을 받은 모델이기 때문에 해당 challenge 대회에서 사용한 데이터 셋을 사용했습니다. 해당 데이터 셋의 간단한 정보는 아래와 같습니다 (ISBI challenge에 대해서는 FCN 관련 글에서도 설명해놨으니 참고해주세요!)
- Dataset: Transmitted light microscopy images (phase contrast and DIC((Differential Interference Contrast)))
- Transmitted light microscopy = 투과 광선 현미경검사
- Phase-contrast microscopy = 위상차 현미경
- Differential interference contrast microscope ( DIC) = 차등간섭대비현미경
- Input Size: 512×512
- Deep Learning Framework: Caffe
- Inference Time: a second on a recent GPU
- Training time: 10 hours → "5. Conclusion"에서 알 수 있는 정보
- GPU: NVidia Titan (6 GB) → "5. Conclusion"에서 알 수 있는 정보
- Dataset: Transmitted light microscopy images (phase contrast and DIC((Differential Interference Contrast)))
1. Introduction

- 기존 CNN에 대해서 짧게 설명하고 있습니다.
- AlexNet 소개
- VGGNet 소개
- 2015년 당시에 CNN 최신 모델은 VGGNet 또는 GoogLeNet이었습니다.

- 기존 CNN은 보통 single classification task에 사용되었습니다.
- 하지만 biomedical image processing 분야에서는 한 이미지 내에 모든 pixel을 classification을 하는 segmentation task가 중요하게 사용되기도 합니다. (예를 들어, 암의 위치 또는 크기를 segmentation 할 필요가 있겠죠?)
- ImageNet에 비해 (biomedical tasks을 위한) biomedical image 수가 충분하지 않다는 문제가 있습니다.
- by reach = 손이 닿지 않는 곳에 = "가능성이 없는" 늬앙스
- 이러한 데이터 수 문제를 해결하기위해 (UNet 이전에) patch 기반의 학습 기법("Ciresan et al"="Sliding-window setup")을 이용해 data 수를 늘리려는 시도를 했습니다.
- Sliding-window setup 방식은 FCN 글에서 설명해 놨으니 참고해주세요.
- Sliding window 기반 딥러닝 segmentation 모델이 EM (Electron Microscopy: 전자현미경) segmentation 종류인 ISBI 2012 challenge 우승했습니다.
(↓↓↓ EM segmentation ↓↓↓)
http://brainiac2.mit.edu/isbi_challenge/home
About the 2D EM segmentation challenge | ISBI Challenge: Segmentation of neuronal structures in EM stacks
brainiac2.mit.edu

- "Ciresan et al"="Sliding-window setup" 방식은 2가지의 문제점이 있는데, 이 부분도 FCN 글을 참조해주시길 바랍니다!

- 이 문단은 FCN에 대한 설명이므로 FCN 글을 참조해주세요!
- successive layers = deconv layers

- "그림1"에서 볼 수 있듯이 expanding path(=upsampling part) 를 보면 기존 FCN 보다 더 많은 conv filter channel들이 사용되었다는걸 확인 할 수 있습니다. 이를 통해 저자는 "propagate context information to higher resolutioin layer" 가 가능하다고 주장하고 있네요.

- "segmentation map only contain ~ in the input image"→ segmentation map (=아래 "그림1"을 보면 최종 output결과를 segmentation map이라고 부름) 에는 입력 영상에서 전체 context를 "available"할 수 있는 pixel만 포함(=segmentation)된다고 합니다. → 이 부분은 그냥 segmentation map 결과가 context (의미 or 객체) 단위로 표현된다는 것으로 이해했습니다. 예를 들어, 아래 그림처럼 입력 이미지는 고양이의 질감이나 다양한 정보들을 확인 할 수 있는데 반해, segmentation map에서는 오로지 context 정보만 확인할 수 있습니다.
- Valid part에 대한 정확한 의미는 잘 모르겠네요. Valid part만 보면 segmentation map을 의미하는거 같다가도, "valid part of each convolution"을 보면 convolution과 연관지어 해석해야 할 것 같기도합니다. Segmentation map에 적용되는 각각의 convolution이라고 해석해야하는건지.....

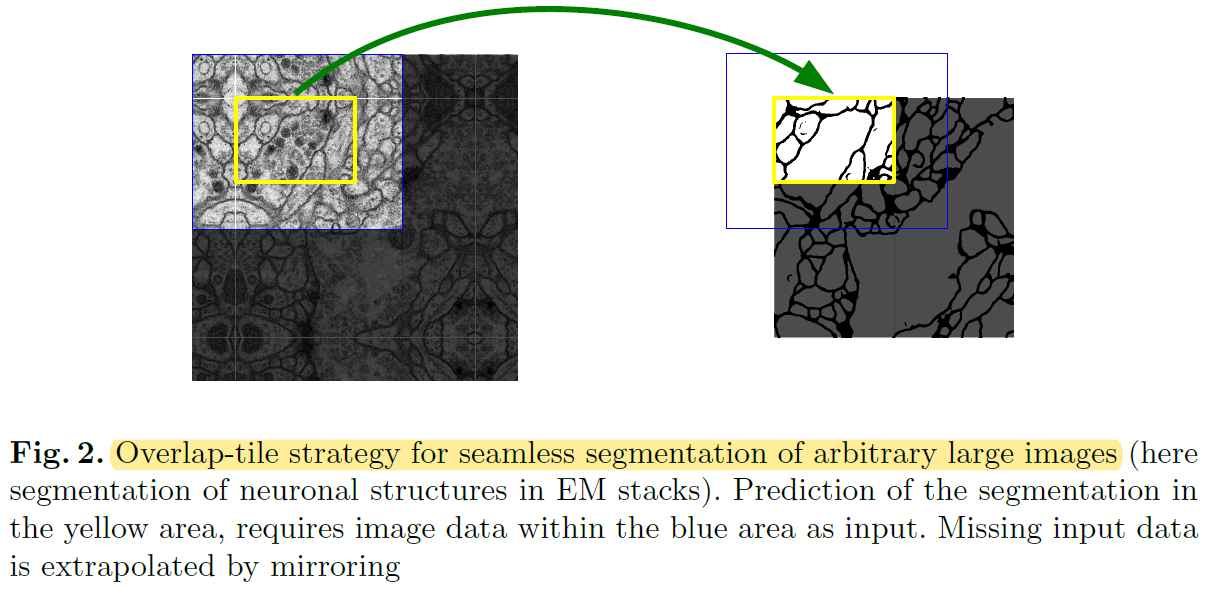
- This strategy(=not using FC layer)는 입력 이미지 크기에 제한 받지 않고 (=arbitrarily large images) segmentation이 가능하게 만들었는데, over-tile strategy를 이용해 좀 더 seamless(=매끄러운) segmentation이 가능하게 만들었습니다.
- 지금부터 아래 "그림2(=over-tile strategy)"에 대해서 좀 더 자세히 설명 해보도록 하겠습니다.
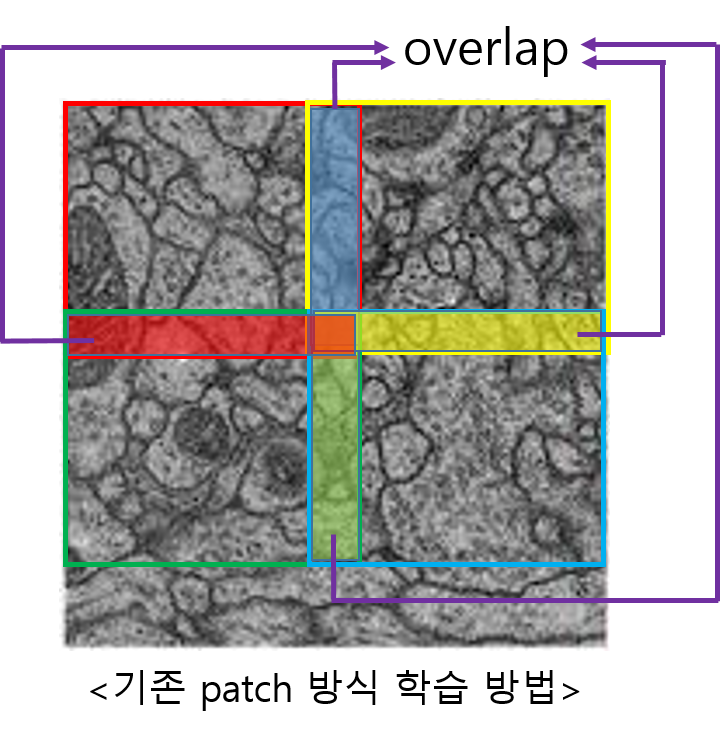
- 아래 "그림3"과 같이 기존의 patch 방식으로 학습하면 overlapping되는 부분이 많이 생깁니다.
- 이러한 문제는 computation의 redundancy를 증가시키기도 합니다.
- Unet에서는 이러한 문제를 해결하기 위해 overlap-tile strategy를 적용시켰습니다.
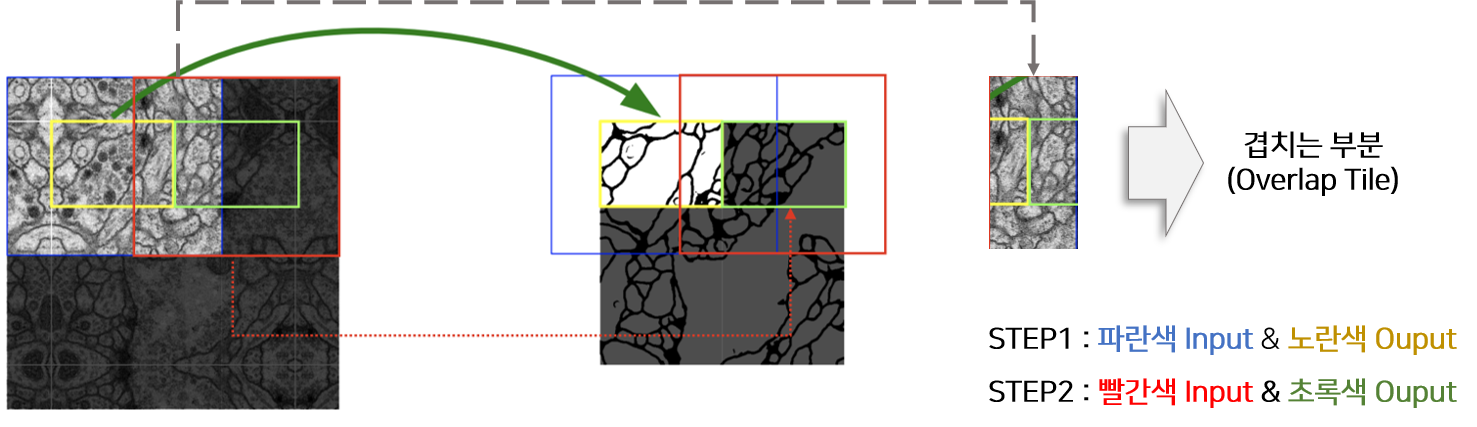
- Over-tile strategy를 설명하기에 앞서 “그림1”의 Unet 구조를 보면 input size와 output size가 다릅니다.
- 다시 말해, 실제 segmentation 되는 부분은 input image의 일부분이라는 말과 같습니다. (→ 아래 "그림6"의 오른쪽 이미지 참고)

- Overlap-tile strategy 을 이용하면 실제 segmentation 되는 부분은 overlapping이 안 되는 것을 알 수 있습니다. (이 부분 때문에 “seamless segmentation”이라는 표현을 쓴 건지....)

- To predict the pixels in the border region of the image, the missing context is extrapolated by mirroring the input image.
- the pixels in the border region of the image → input image의 주황색 bounding box(=the border region) 내부(in) pixel들
- 실제 segmentation되는 부분(=오른쪽 그림에서 “원본 이미지“)이 ground truth의 크기이기 때문에, input으로 들어가는 이미지에 추가 작업을 진행해줘야 합니다.
- 이 논문에서는 input size에 해당하는 빨간색 bounding box와 실제 segmentation size에 해당하는 주황색 bounding box를 뺀 나머지 부분을 “missing context”라 언급하고, mirroring 기법을 이용해 missing context 부분을 extrapolation(=추론) 하여 input image를 만들어 준다고 합니다.

- 이러한 over-tile strategy를 이용해서 input image resolution에 제한받지 않고 segmentation 학습이 가능해졌다고 말하고 있습니다.

- Segmentation 학습을 위해 제한적인 training dataset을 갖고 있었기 때문에 excessive data augmentation을 진행해 학습 데이터 수를 증가시켰다고 합니다.
- 여기에서는 elastic deformation augmentation 방식을 설명했지만 "3.1. Data augmentation" 파트에서 실제로 학습에 적용된 다양한 augmentation 방식들을 소개하고 있기 때문에 추가적인 설명은 "3.1. Data augmentation" 파트에서 하도록 하겠습니다.
- 최근에는 "As for our tasks ~ the available training images"라는 말이 상식적이라 최근 논문에는 굳이 사용하지 않지만, 이 당시 (2015) 까지만 하더라도 data augmentation에 대한 언급을 한 문단으로 따로 해줬나보네요.

- biomedical image segmentation (ex: cell segmentation) 같은 경우는 인접한 세포들간을 분리하는 segmentation을 적용하는 것이 쉽지 않습니다. (→ Instance segmentation)
(↓↓↓ Instance segmentation VS Semantic segmentation↓↓↓)
https://89douner.tistory.com/113
1. Segmentation이 뭔가요?
안녕하세요~ 이번 Chapter에서는 segmentation에 대해서 알아보도록 할거에요. Segmentation을 하는 방식에는 여러가지가 있지만, 이번 chapter에서는 딥러닝을 기반으로한 segmentation에 대해서 알아보도록
89douner.tistory.com
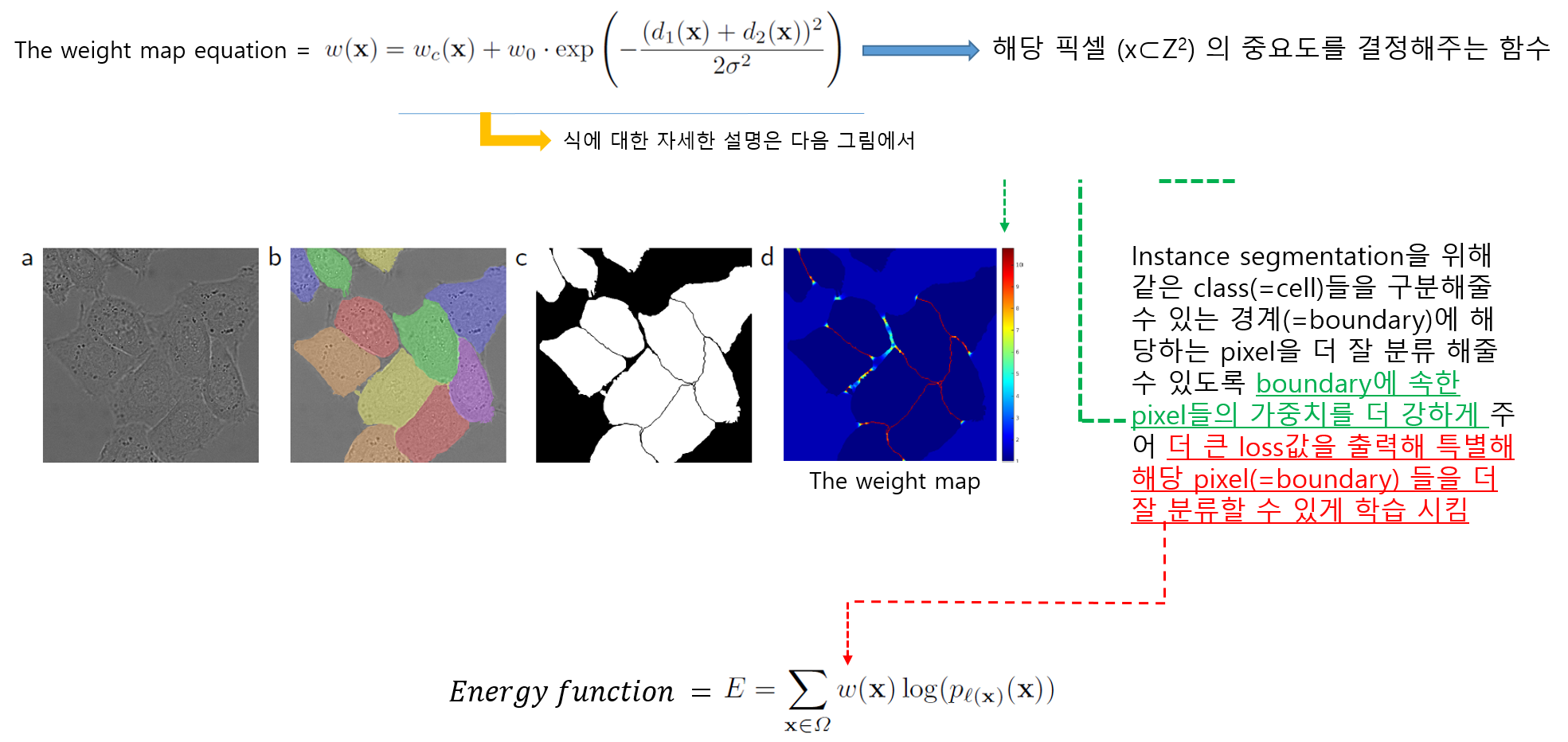
- 이 논문에서는 인접한 cell들 사이에 경계가 될 수 있는 background (=the separting background labels) 를 잘 segmentation 해줄 수 있도록 별도의 loss function을 사용했다고 합니다 (해당 부분은 "3.Training" 파트에서 자세히 설명하도록 하겠습니다)
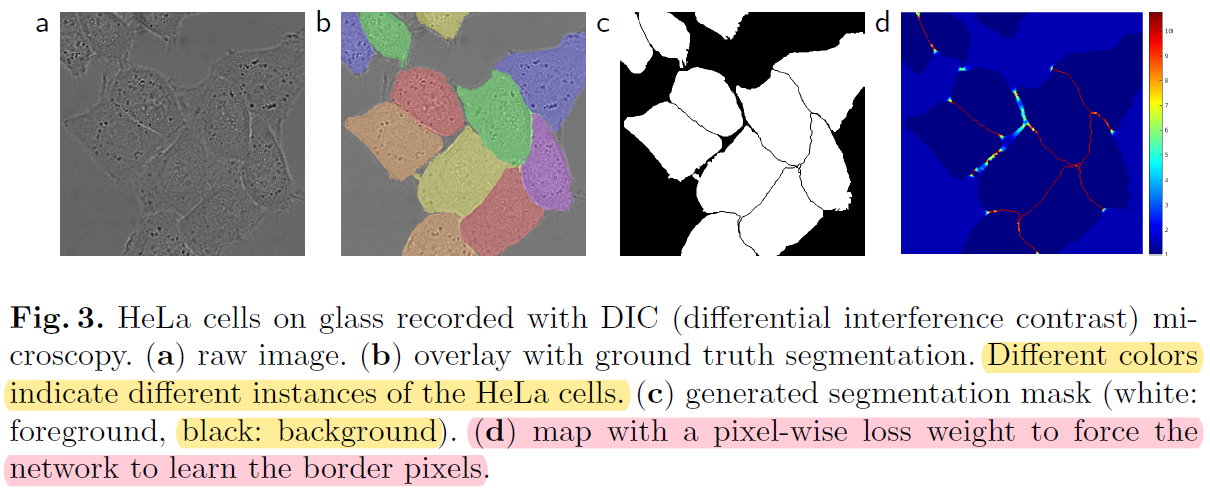

- UNet은 2D transmitted light datasets을 기반으로하는 (대표적인) 두 challenging 대회에서 기존 모델들과 큰 격차(=by a large margin)로 우승을 했다고 합니다. (Transmitted light microscopy = 투과 광선 현미경검사)
- A 대회: Segmentation of neuronal structures in EM (Electron Microscopy: 전자현미경) stacks at ISBI 2012
- B 대회: Light microscopy (=광선 현미경검사) images from the ISBI cell tracking challenge 2015
2. Network Architecture

- UNet 구조는 앞에서 추상적으로 설명했기 때문에 이 부분에서는 좀 더 구체적으로 설명하도록 하겠습니다.
- Contracting path
- Two 3×3 Conv filter (stride=1) with zero padding of input feature map → feature map size-reduction → 2×2 max-pooling → feature map size-reduction (to Half)
- 이 과정을 반복해서 적용
- Expansive path (Expansive path는 크게 세 단계로 나눌 수 있습니다.)
- 2x2 De-Conv filter that halves the number of feature channels. (아래 "그림10"에서 보라색 점선 화살표를 보면 input channel (=512)을 256으로 줄여주는걸 확인할 수 있습니다.)
- Copy and Crop a → "concatenation with the correspondingly cropped feature map from the contracting path"
- 아래 "그림10"에서 ①②라고 되어 있는 부분을 기준으로 설명하겠습니다.
- Crop: Contracting path에 있는 136×136 feature map size → crop → 104×104 feature map size
- 정확한건 아니지만 이 과정에서 앞서 언급한 mirroring이 적용된 부분이 제거 되는 것으로 추론됩니다. (어떻게 crop하는지 코드를 살펴봐야겠네요)
- Copy and Concatentation: Contracting path에서 crop된 (104×104)×256 feature map을 Copy → 아래 "그림10" 기준으로 같은 층에 존재하는 expansive path의 (104×104)×256 feature map에 concatenation → Concatenation 결과: (104×104)×512 feature map 생성
- Final layer: 388×388×64 feature map을 output segmentation map으로 만들어주기 위해, 1×1 conv filter를 이용해 388×388×2 feature map으로 만들어줌 (=mapping)
- 아래 "그림10"을 기준으로 보면 2는 classification해야 할 class 개수인 것 (binary classification: cell인지 아닌지(=background)) 같은데 아니라면 댓글 달아주시면 감사하겠습니다!
- Total 23 convolutional layers = 18 conv 3x3 (blue arrows) + 4 up-conv 2x2 (green arrows) + 1 final layer (an orange arrow) = 23
- Contracting path


- 2×2 max-pooling을 적용하기 위해서 conv layer에 입력으로 들어오는 feature map size가 x, y축 모두 짝수가 되도록 고려해서, input title size를 설정해줘야 over-tile strategy가 잘 적용될 수 있다는 것을 언급하는 듯 합니다.
3. Training

- Optimizer
- Stochastic Gradient Descent (SGD)
- Momentum: 0.99
- Deep learning framework: Caffe
- Batch: A single image
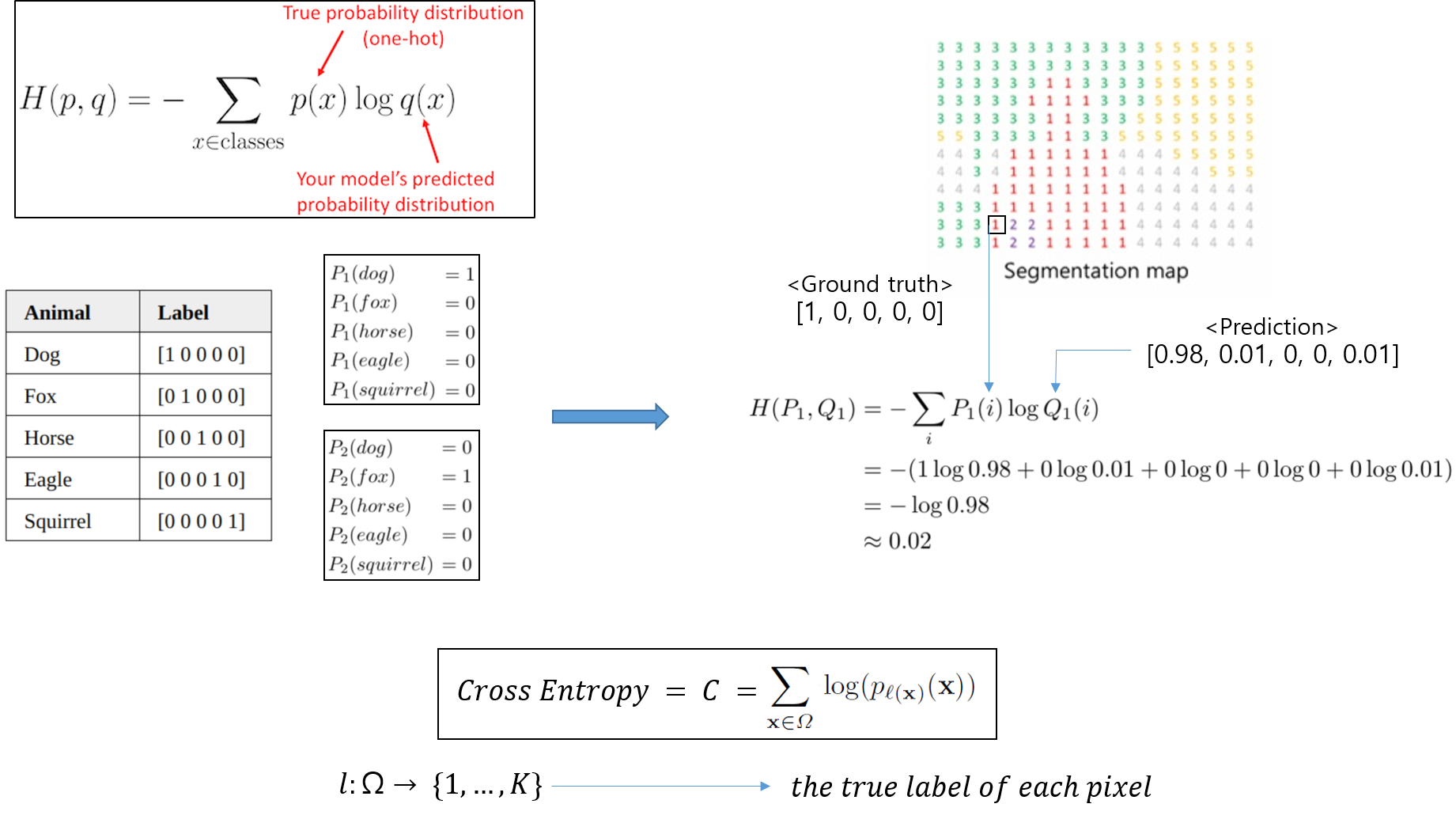
1) Loss function
- UNet은 아래와 같은 loss function을 사용하고 있습니다.
- 논문에서 글로 설명하는 내용들을 그림으로 대체해서 설명해보도록 하겠습니다.

1-1) SoftMax

1-2) Cross-Entropy
1-3) Weight Map
- 먼저 weight map equation 식 자체에 대한 concept 부터 이해하도록 하겠습니다.
- 다음은 \(w_{c}(x)\) 수식을 설명하겠습니다.
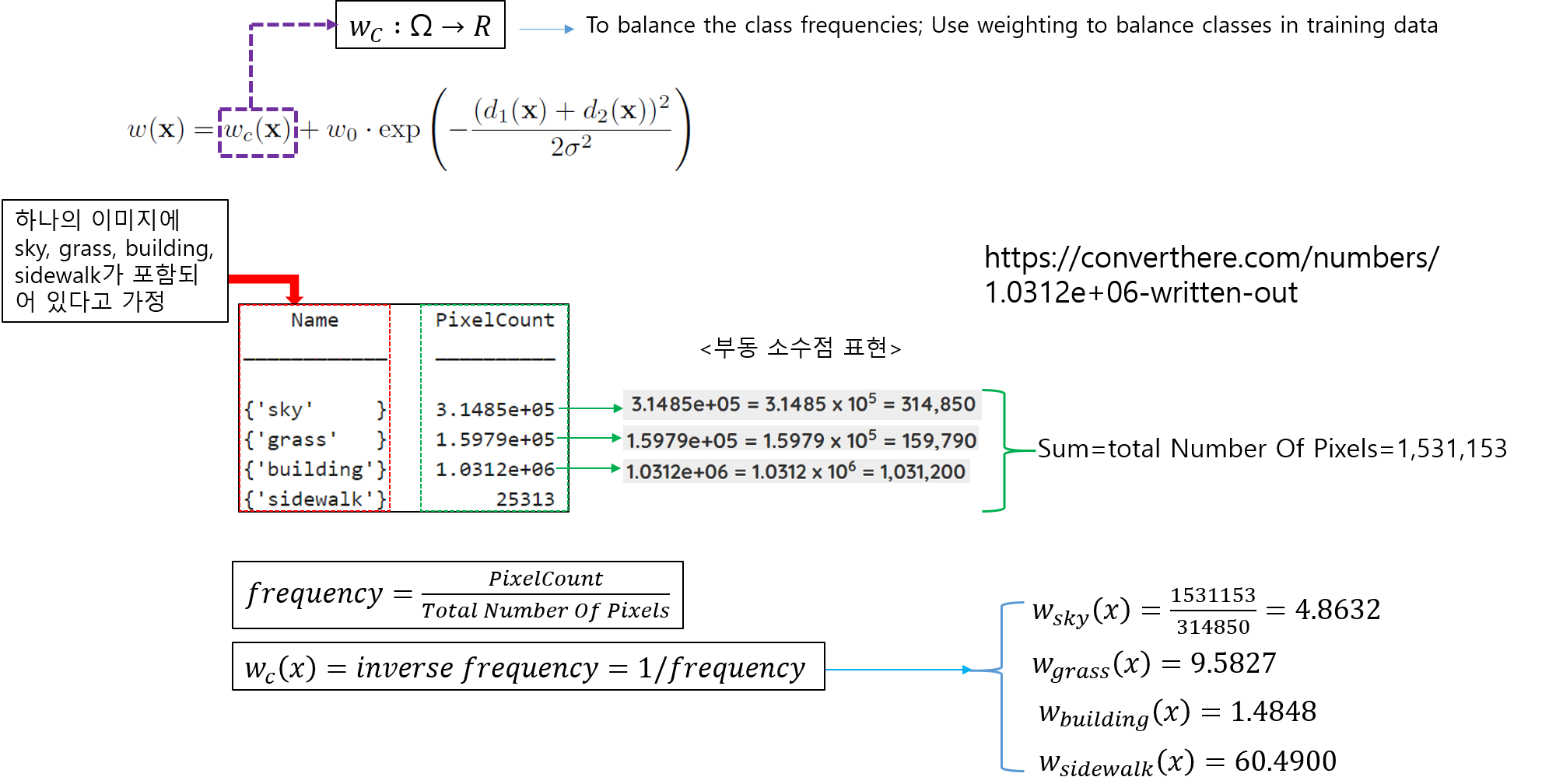
(↓↓↓\(w_{c}(x)\) 수식 관련 reference site↓↓↓)
https://kr.mathworks.com/help/vision/ref/nnet.cnn.layer.pixelclassificationlayer.html
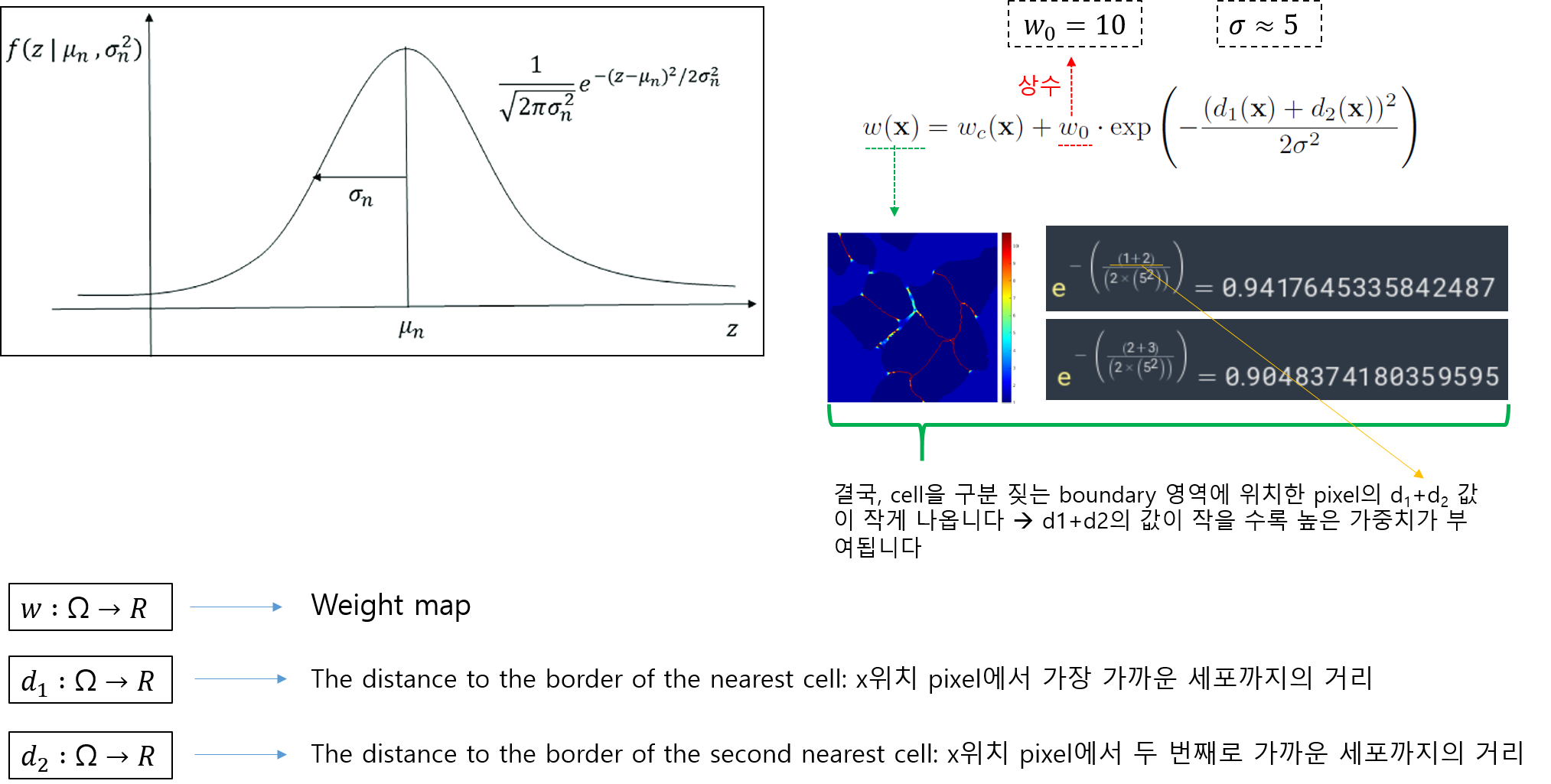
- weight map equation에서 exp 수식 부분 설명
- weight map equation 수식의 오른쪽 수식은 guassian distribution과 유사합니다.
- 개인적으로 gaussian function 수식과 weight map equation 수식의 오른쪽 수식이 어떤 관계가 있을 거라 생각 하는데, 그 이유는 이 논문에서는 σ 개념을 도입했기 때문입니다. 하지만, 좀 더 구체적인 설명은 하지 않고 있어서 해당 exp 수식이 정말 guassian function 개념을 기반으로 설명 될 수 있는지 잘 모르겠네요... (exp 수식을 왜 사용했는지 좀 더 알고 싶은데.... 시간이... 혹시 아시는 분 있으면 답글 달아주세요!)
2) Weight initialization

- Gaussian distribution initialization (=standard deviation distribution)
- Standard deviation: \(\sqrt{\frac{2}{N}}\)
- N = conv filter size × conv filter channel
3.1 Data Augmentation

- 이 논문에서 언급하고 있는 data augmentation 방식은 총 가지 입니다.
- Shift
- Rotation
- Gray value
- Random elastic deformation
- Random elasstic deformation에 대해서 다양하게 설명하고 있는데, 해당 설명은 아래 그림으로 대체하도록 하겠습니다.

4. Experiments
이 부분에서는 UNet을 적용한 세 가지 task (3가지 dataset 종류)에 대해서 소개합니다. 세 가지 task에 대한 설명은 그냥 논문에 있던 글들을 요약해서 정리해 놓았습니다. 대신에 성능평가를 위해 사용한 evaluation 종류에 대해 자세히 설명하도록 해보겠습니다.

- 이 논문에서 UNet 모델을 이용해 3가지 segmentation task에 적용해봤다고 합니다.
- First task: The segmentation of neuronal structures in electron microscopic (EM) recordings
- Dataset: the EM segmentation challenge (at ISBI 2012)
- 20 training images "512×512" (from serial section transmission electron microscopy of the Drosophila first instar larva ventral nerve cord (VNC))
- Drosophila: 초파리
- Instar larval: 어린 유충
- Ventral nerve cord: 복부 신경 코드
- Other tasks: A cell segmentation task in light microscopic images (=A part of the ISBI cell tracking challenge 2014 and 2015)
- Second task
- Dataset: PhC-U373 containing Glioblastoma-astrocytoma U373 cells on polyacrylimide substrate (from phase-contrast microscopy)
- Glioblastoma-astrocytoma U373 cells on polyacrylimide substrate → 생물학 용어인거 같은데 잘 모르겠네요;;
- 35 partially annotated training images
- Dataset: PhC-U373 containing Glioblastoma-astrocytoma U373 cells on polyacrylimide substrate (from phase-contrast microscopy)
- Third task
- Dataset: DIC-HeLa (from differential interference contrast (DIC) microscopy)
- 20 partially annotated training images
- Second task
- First task: The segmentation of neuronal structures in electron microscopic (EM) recordings

1) Pixel error
- Pixel error는 prediction과 ground truth에 pixel마다 할당된 class들끼리 불일치하는 pixel들의 개수가 전체 pixel들을 기준으로 어느정도 비율이 되는지 알려주는 metric입니다.
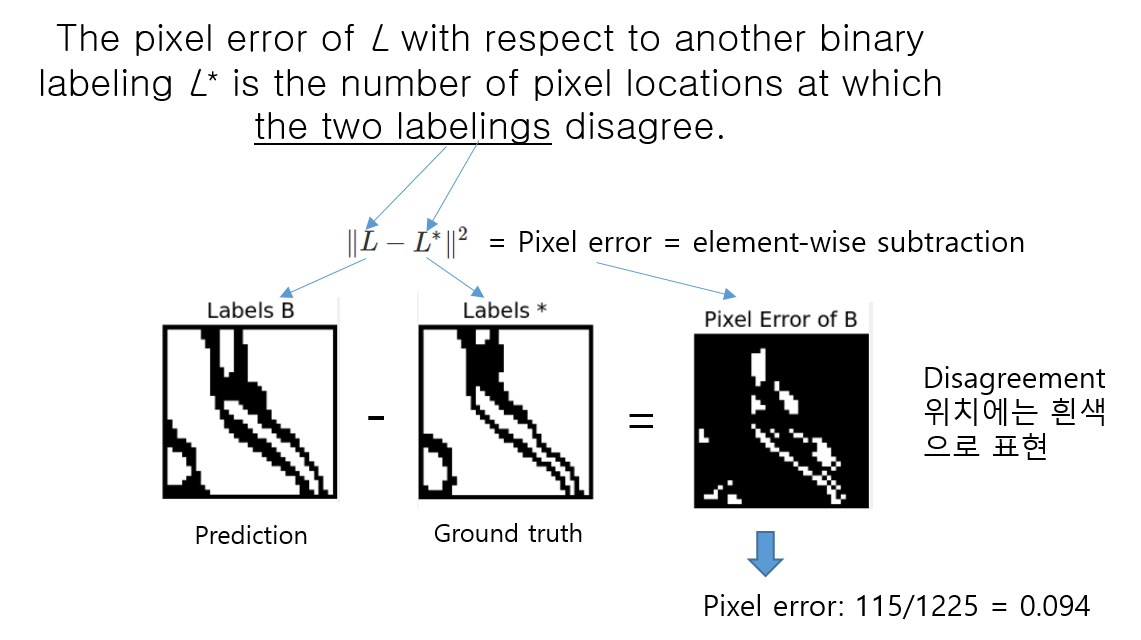
- Warping error와 Rand error는 digital topology error과 관련이 있는듯 합니다. 이 부분은 topology(위상수학)에 대한 이해를 기반으로 해석해야 하는데, 시간상 해당 부분을 살펴보지 못했습니다. 좀 더 디테일한 설명을 원하신다면 아래 reference를 참고해주시고, 추후에 시간이나면 설명해 놓도록 하겠습니다!
(↓↓↓Evaluation 관련 reference site↓↓↓)
https://imagej.net/plugins/tws/topology-preserving-warping-error
Topology preserving warping error
The ImageJ wiki is a community-edited knowledge base on topics relating to ImageJ, a public domain program for processing and analyzing scientific images, and its ecosystem of derivatives and variants, including ImageJ2, Fiji, and others.
imagej.github.io
'Deep Learning for Computer Vision > Segmentation' 카테고리의 다른 글
| 2. FCN (1) | 2021.07.08 |
|---|---|
| 1. Segmentation이 뭔가요? (1) | 2020.02.17 |